InstaDeep推出Nucleotide Transformer v3 (NTv3):一个专为单核苷酸分辨率的1 Mb上下文长度设计的新多物种基因组学基础模型。
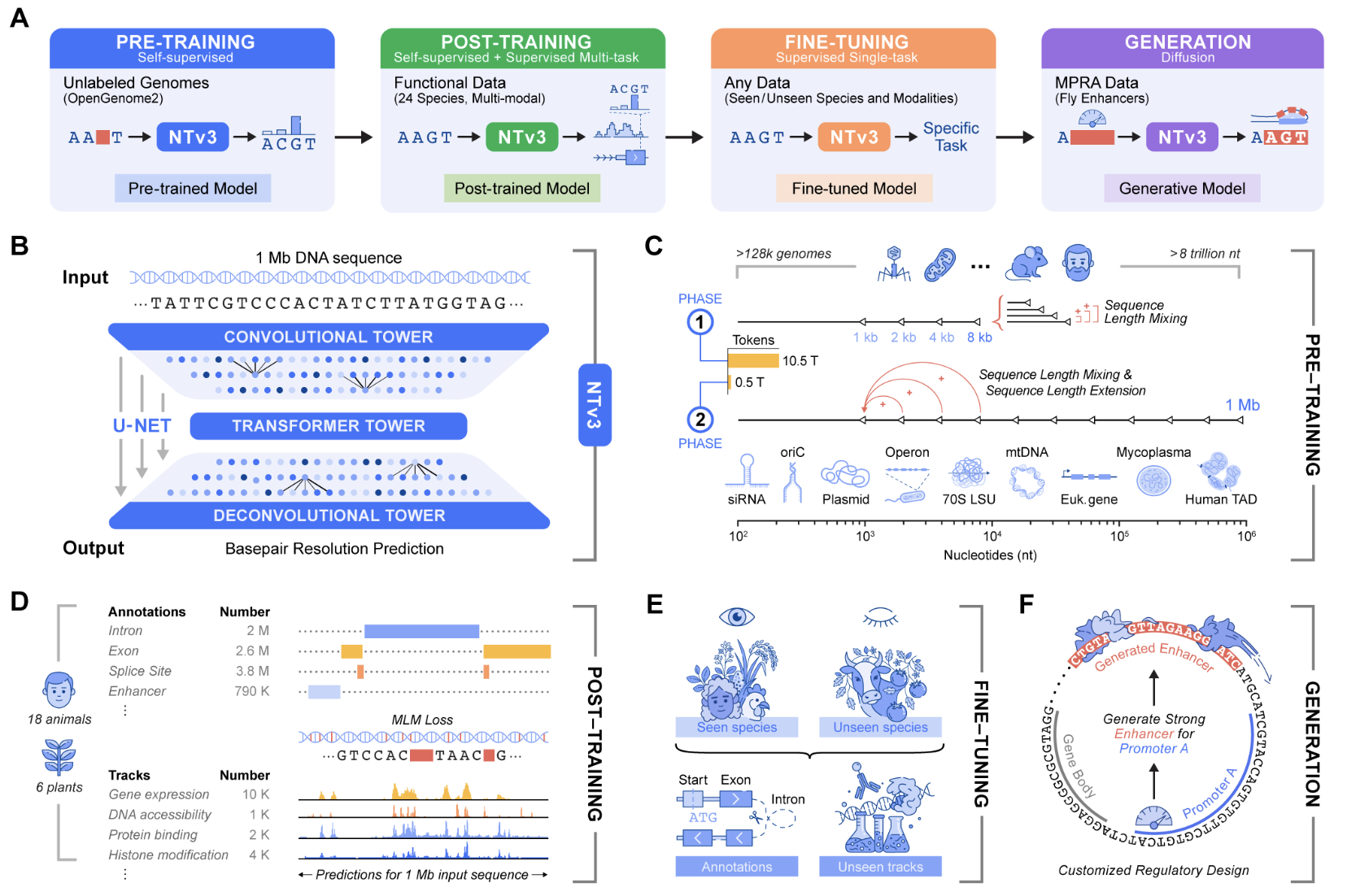
基因组预测和设计现在需要连接局部基序与百万碱基尺度调控语境,并且能够在多种生物体上运行的模型。Nucleotide Transformer v3,简称NTv3,是InstaDeep为此设置推出的新多物种基因组基础模型。它将表示学习、功能轨迹和基因组注释预测以及可控序列生成统一在一次主骨架上,该骨架在1 Mb语境中按单核苷酸分辨率运行。
早期的Nucleotide Transformer模型已经表明,在成千上万的基因组上进行自监督预训练,可以为分子表型预测提供强大的特征。原始系列包括了从5亿到25亿参数的模型,这些模型在3200个人类基因组以及来自多种物种的850个额外基因组上进行了训练。NTv3保留了这一序列仅预训练的思路,并将其扩展到更长语境,并添加了显式功能性监督和生成模式。
https://huggingface.co/spaces/InstaDeepAI/ntv3
1 Mb基因组窗口的架构
NTv3使用了一种U-Net风格的架构,旨在针对非常长的基因组窗口。一个卷积下采样塔压缩输入序列,一个Transformer堆栈在该压缩空间中模型长距离依赖关系,一个反卷积塔恢复基础分辨率以进行预测和生成。输入使用字符级在A、T、C、G、N上进行标记,并使用特殊标记,如<unk>、<pad>、<mask>、<cls>、<eos>和<bos>。序列长度必须是128个标记的倍数,参考实现使用填充来强制执行此约束。所有公共检查点都使用单碱基标记化,标记词汇大小为11个标记。
最小的公共模型NTv3 8M pre大约有7.69M个参数,隐藏维度256,FFN维度1,024,2个Transformer层,8个注意力头,和7个下采样阶段。在高端,NTv3 650M使用隐藏维度1,536,FFN维度6,144,12个Transformer层,24个注意力头,和7个下采样阶段,并为物种特定预测头部添加了条件层。
训练数据
NTv3模型是在OpenGenome2资源的9万亿碱基对上使用基础分辨率遮蔽语言模型进行预训练的。在此阶段之后,该模型与一个联合目标进行后训练,该目标结合了持续的自我监督以及在大约16,000个功能轨迹和来自24种动物和植物物种的注释标签上的监督学习。
性能和Ntv3基准
在经过后训练后,NTv3在跨物种的功能轨迹预测和基因组注释方面实现了最先进的准确性。它在现有公共基准上优于强大的序列到功能模型和之前的基因组基础模型,并在新定义的Ntv3基准上也表现出色,该基准是一种受控下游微调套件,具有标准化的32 kb输入窗口和基础分辨率输出。
Ntv3基准目前包括106个长时间、单核苷酸、交叉测定、跨物种的任务。因为NTv3在24种物种的数千条轨迹上进行了后训练,因此模型学习了一个共享的调控语法,可以在生物体和测定之间迁移并支持协调一致的长距离基因组到功能的推断。
从预测到可控序列生成
除了预测之外,NTv3可以通过遮蔽扩散语言模型微调为一个可控生成模型。在此模式下,模型会接收编码所需增强子活度水平和启动子选择性的条件信号,并以与这些条件一致的方式来填充DNA序列中遮蔽的跨度。
在启动材料中描述的实验中,团队设计了1,000个具有指定活度和启动子特异性的增强子序列,并与Stark实验室合作在体外使用STARR seq测定进行验证。结果表明,这些生成的增强子在STARR seq测定中恢复了预期的活度排序,并与基线相比提高了超过2倍的启动子特异性。
比较表
| 尺寸 | NTv3 (Nucleotide Transformer v3) | GENA-LM |
|---|---|---|
| 主要目标 | 统一的多物种基因组基础模型,用于表示学习、序列到功能预测和可控序列生成 | 用于许多监督基因组预测任务的DNA语言模型系列,专注于转移学习 |
| 架构 | U-Net风格的卷积塔、Transformer堆栈、反卷积塔、单碱基分辨率语言模型,后训练版本添加了多物种条件和任务特定头部 | 基于BERT的编码模型,有12或24层,以及BigBird变体,具有稀疏注意力,进一步扩展到数百万个碱基对的长期上下文医疗器械 |
| 参数规模 | 模型家族包括8M、100M和650M参数 | 基础模型有110M参数,大型模型有336M参数,包括110M参数的BigBird变体 |
| 本地语境长度 | 预训练和后训练模型都支持高达1 Mb输入,分辨率为单核苷酸 | BERT模型大约4500个碱基对,使用512个BPE标记,BigBird模型最多36000个碱基对,使用4096个标记 |
| 语境扩展机机制 | 在Transformer层之前使用U-Net风格的卷积塔聚合长期上下文,同时保持单碱基分辨率;在发布的检查点中,语境长度固定为1 Mb | 在BigBird变体中使用稀疏注意力,加上递归记忆Transformer以扩展有效语境到数十万个碱基对 |
| 标记化 | 在A、T、C、G、N和特殊标记上进行字符级标记化;每个核苷酸都是一个标记 | DNA上的BPE标记器,映射到大约4500个碱基对的512个标记;使用两种标记器,一种仅用于T2T,另一种用于T2T加上1000G SNPs加上多物种数据 |
| 预训练语料库大小 | 第一阶段在OpenGenome2上预训练,使用了来自超过128000个物种的约9万亿碱基对 | 仅人类模型在预处理的人类T2T v2加上1000 Genomes SNPs上训练,大约有480 × 10^9个碱基对,多物种模型在人类和多物种数据上联合训练,大约有1072 × 10^9个碱基对 |
| 物种覆盖率 | 在OpenGenome2预训练和多物种监督提供中超过128000种,24种动物和植物物种的监督学习 | 以人类为重点的模型以及酵母、拟南芥和果蝇的物种特定模型和多物种模型来自ENSEMBL基因组 Username:Password: |
| 监督后训练信号 | 大约有16000个跨越大约10种测定类型和大约2700个组织的24种物种的功能轨迹,用于对骨干进行条件化以及训练功能头部 | 在多个监督任务上微调,包括启动子、剪接点、果蝇增强子、染色质图谱和polyadenylation位点,在LM顶部有特定任务的头部 |
| 生成能力 | 可以通过遮蔽扩散语言模型微调为可控生成模型,用于设计具有指定活度和启动子选择性的增强子序列,并使用STARR seq测定进行实验验证以确认预期活度顺序和改善的启动子特异性 | 主要用作遮蔽语言模型和特征提取器,支持通过MLM进行序列完成,但主要出版物侧重于预测任务而不是显式可控序列设计 |
主要结论
- NTv3是一个长距离、多物种基因组基础模型:它将表示学习、功能轨迹预测、基因组注释和可控序列生成统一在一个U Net风格的架构中,该架构支持24种动物和植物物种的1 Mb核苷酸分辨率语境。
- 该模型在9万亿碱基对上进行训练,具有联合自我监督和监督目标:NTv3在9万亿碱基对上使用基础分辨率遮蔽语言模型进行预训练,然后使用超过16,000个功能轨迹和来自24种物种的注释标签的后训练,这些注释标签使用了混合持续自我监督和监督学习的联合目标。
- NTv3在Ntv3基准上实现了最先进的性能:后训练后,NTv3在跨物种的功能轨迹预测和基因组注释方面达到了最先进的准确性,并在公共基准和Ntv3基准上优于之前的序列到功能模型和基因组基础模型,Ntv3基准包含106个具有32 kb输入和基础分辨率输出的标准化长距离下游任务。
- 相同的骨干支持与STARR seq验证的增强子设计:NTv3可以通过使用遮蔽扩散语言模型微调为可控生成模型,用于设计具有指定活度和启动子选择性的增强子序列,这些设计通过STARR seq测定进行实验验证以确认预计活度顺序和改善的启动子特异性。